Statystyka lokalna I Morana
Lokalna wersja statystyki Morana jest najbardziej popularną analizą określaną jako LISA (Local Indicators of Spatial Association) (Luc Anselin 19951)). W odróżnieniu od globalnej statystyki Morana wyznacza ona lokalną autokorelację przestrzenną, a zatem określa podobieństwo jednostki przestrzennej wobec sąsiadów i bada istotność statystyczną tej zależności.
Lokalny współczynnik autokorelacji Morana
Lokalna postać współczynnika  Morana dla obserwacji
Morana dla obserwacji  określona jest wzorem:
określona jest wzorem:
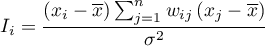
gdzie:
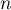 – liczba obiektów przestrzennych (liczba punktów lub wielokątów),
– liczba obiektów przestrzennych (liczba punktów lub wielokątów),
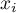 ,
, 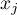 – to wartości zmiennej dla porównywanych obiektów,
– to wartości zmiennej dla porównywanych obiektów,
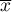 – to średnia wartość zmiennej dla wszystkich obiektów,
– to średnia wartość zmiennej dla wszystkich obiektów,
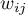 – elementy przestrzennej macierzy wag (zalecana jest macierz standaryzowana rzędami do jedynki),
– elementy przestrzennej macierzy wag (zalecana jest macierz standaryzowana rzędami do jedynki),
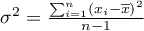 – wariancja
– wariancja
Interpretacja lokalnego współczynnika Morana jest analogiczna do jego globalnego odpowiednika jednak w znacznym stopniu zależy od wybranej macierzy wag. Najczęściej wagi niezerowe są przypisywane tylko do obiektów sąsiadujących, w rezultacie współczynnik lokalny określa podobieństwo jedynie obiektów znajdujących się w strefie sąsiedztwa. Standaryzacja rzędami do jedynki ułatwia natomiast porównywanie wartości współczynników uzyskanych dla różnych obiektów, gdyż wartość oczekiwana dla każdego współczynnika jest wówczas taka sama.
Wysokie wartości współczynnika wskazują na występowanie klasterów podobnych wartości, niskie - na występowanie tzw. hot spots, a wartości bliskie wartości oczekiwanej 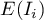 na losowy rozkład badanej zmiennej w przestrzeni.
na losowy rozkład badanej zmiennej w przestrzeni.
Wartość oczekiwana określona jest wzorem:
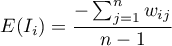
Istotność współczynnika autokorelacji Morana
Testując istotność statystyczną związku między sąsiadującymi obiektami bada się hipotezy:
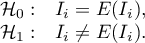
Statystyka testowa ma postać:
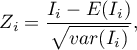
gdzie:
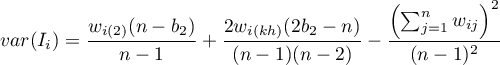 – wariancja w rozkładzie losowym,
– wariancja w rozkładzie losowym,
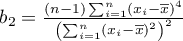 ,
,
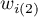 – suma kwadratu wag dla wiersza
– suma kwadratu wag dla wiersza  ,
,
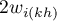 - suma możliwych iloczynów wag dla wiersza
- suma możliwych iloczynów wag dla wiersza  po wykluczeniu iloczynów o tych samych indeksach.
po wykluczeniu iloczynów o tych samych indeksach.
Statystyka 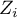 ma asymptotycznie (dla dużych liczności) rozkład normalny.
ma asymptotycznie (dla dużych liczności) rozkład normalny.
Wartość 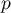 , wyznaczoną na podstawie statystyki testowej, porównujemy z poziomem istotności
, wyznaczoną na podstawie statystyki testowej, porównujemy z poziomem istotności 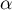 :
:
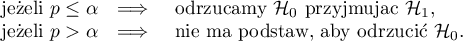
Ze względu na problem braku niezależności współczynników wyliczanych dla sąsiednich obiektów sugeruje się stosowanie skorygowanego poziomu istotności 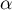 . Proponowane poprawki to: poprawka Bonferroniego:
. Proponowane poprawki to: poprawka Bonferroniego: 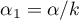 lub Sidaka:
lub Sidaka: 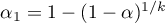 , gdzie
, gdzie 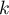 jest średnią liczbą sąsiadów.
jest średnią liczbą sąsiadów.
Warstwy mapy
Kombinacja informacji z wykresu punktowego Morana (podział obiektów na High-High, Low-Low, Low-High, High-Low) i z istotności statystyki lokalnej Morana przedstawia na mapie tzw. reżimy przestrzenne:
- Istotne statystycznie obiekty High-High (obiekty o wysokich wartościach otoczone przez obiekty o wysokich wartościach) zaznaczone są na mapie kolorem czerwonym;
- Istotne statystycznie obiekty Low-Low (obiekty o niskich wartościach otoczone przez obiekty o niskich wartościach) zaznaczone są na mapie kolorem niebieskim;
- Istotne statystycznie obiekty Low-High (obiekty o niskich wartościach otoczone przez obiekty o wysokich wartościach) zaznaczone są na mapie kolorem jasno-niebieskim;
- Istotne statystycznie obiekty High-Low (obiekty o wysokich wartościach otoczone przez obiekty o niskich wartościach) zaznaczone są na mapie kolorem jasno-czerwonym.
Okno z ustawieniami opcji lokalnej analizy Morana wywołujemy poprzez menu Analiza przestrzenna → Statystyki przestrzenne → Statystyka lokalna I Morana.
Przykład c.d. (katalog: leukemia, plik: leukemia)
Analizie poddamy dane dotyczące białaczki.
- Mapa
leukemiazawiera informacje o lokalizacji 281 wielokątów (regionów spisowych) w północnej części stanu New York. - Dane do mapy
leukemia:- Kolumna
CASES– liczba przypadków białaczki w latach 1978-1982 przypisana do poszczególnych obiektów (regionów spisowych). Wartość ta powinna być liczbą całkowitą, tu jednak, zgodnie z opisem Wallera (1994) część przypadków, która nie mogła zostać obiektywnie przypisana do konkretnego regionu, została podzielona proporcjonalnie. Stąd liczności przypadków przypisanych do 281 obiektów nie są liczbami całkowitymi. - Kolumna
POP– liczność populacji w poszczególnych obiektach. - Kolumna
prev– współczynnik częstości występowania białaczki na 100000 osób, dla każdego obiektu w jednym roku: prev=(CASES/POP)*100000/5
Analiza globalna nie dała jednoznacznego rozstrzygnięcia co do występowania autokorelacji przestrzennej. Sprawdzimy więc, czy uda się znaleźć regiony, gdzie częstość występowania białaczki jest nieprzeciętnie wyższa.
By zlokalizować skupiska białaczki oraz regiony kontrastujące z otoczeniem pod względem częstości występowania tej choroby, wyliczymy lokalny współczynnik Morana. Do analizy wykorzystamy zmienną prev oraz proponowaną przez program macierz sąsiedztwa według wspólnej granicy – Queen, standaryzowaną rzędami (by wykorzystać inną macierz należy ją najpierw wygenerować- patrz rozdział: Macierz wag przestrzennych). Wybieramy również jedną z poprawek poziomu istotności.

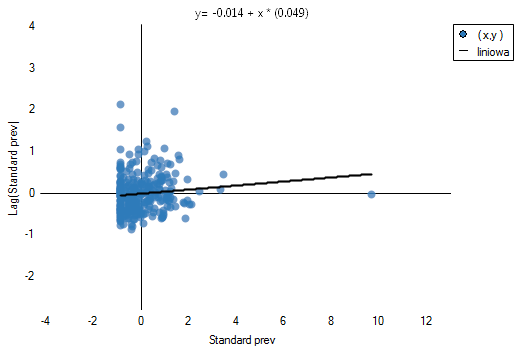
Uzyskany raport przedstawia wartości lokalnych współczynników, wartości statystyki testowej oraz odpowiadające im wartości prawdopodobieństwa testowego. Znajdziemy tu również informacje o ilości rejonów wyznaczających reżimy przestrzenne (High-High, Low-Low, Low-High, High-Low).
Do analizy przypisany jest także wynik, który możemy wyrysować na mapie (przycisk  ) - są to reżimy przestrzenne opisane w raporcie poprzez kolumnę kolor.
) - są to reżimy przestrzenne opisane w raporcie poprzez kolumnę kolor.
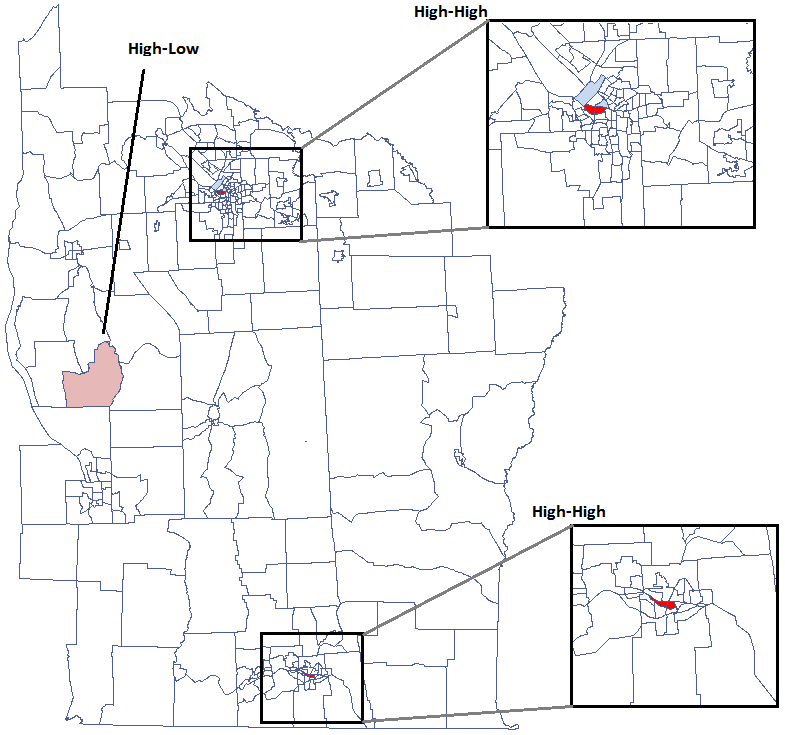
Udało się zlokalizować niewielkie ale istotne skupiska gdzie częstość występowania białaczki jest wyższa. Kolorem czerwonym oznaczone są 2 skupiska (4 regiony spisowe) leżące w mniejszych i bardziej zaludnionych regionach - są to centra klasterów wysokich wartości białaczki. Kolorem jasno-czerwonym oznaczony jest 1 region spisowy o wysokich wartościach współczynnika określającego częstość zachorowania na białaczkę. Region ten jest regionem kontrastującym wobec sąsiednich regionów spisowych, które charakteryzują się stosunkowo niskim współczynnikiem.
Uzyskane wyniki możemy dodatkowo zobrazować kolorując mapę wartościami lokalnego współczynnika Morana 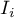 lub też wartościami statystyki testowej bądź wartościami
lub też wartościami statystyki testowej bądź wartościami 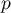 . Wystarczy jedynie wcześniej przekopiować odpowiednie kolumny z raportu do arkusza danych. W tym przykładzie do kolorowania wykorzystamy wartości statystyki testowej
. Wystarczy jedynie wcześniej przekopiować odpowiednie kolumny z raportu do arkusza danych. W tym przykładzie do kolorowania wykorzystamy wartości statystyki testowej 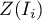 . Po wklejeniu jej do pustej kolumny arkusza danych, w menadżerze map kolorujemy mapę bazową zgodnie z wartościami tej kolumny wybierając odchylenie standardowe o współczynniku 3 jako sposób gradacji kolorów. Dodatnie i wysokie wartości statystyki
. Po wklejeniu jej do pustej kolumny arkusza danych, w menadżerze map kolorujemy mapę bazową zgodnie z wartościami tej kolumny wybierając odchylenie standardowe o współczynniku 3 jako sposób gradacji kolorów. Dodatnie i wysokie wartości statystyki 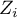 wskazują na występowanie klasterów podobnych wartości, ujemne i niskie - na występowanie tzw. hot spots. Wartości bliskie 0 wskazują natomiast na losowy rozkład badanej wartości w przestrzeni.
wskazują na występowanie klasterów podobnych wartości, ujemne i niskie - na występowanie tzw. hot spots. Wartości bliskie 0 wskazują natomiast na losowy rozkład badanej wartości w przestrzeni.
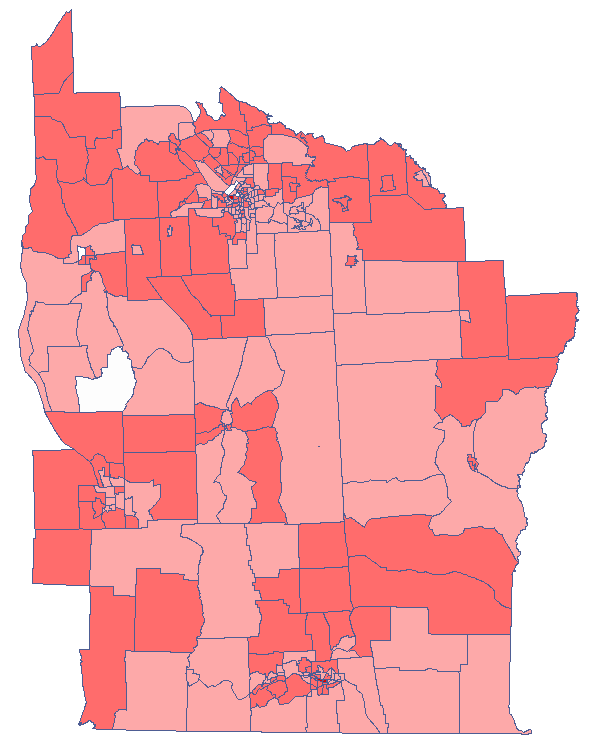
Analizując wygładzoną zmienną prev wzmacniamy efekt klasteryzacji. Uzyskujemy podobny rezultat, ale tym razem lokalizujemy 3 skupiska (19 regionów spisowych) będące centrami klasterów.